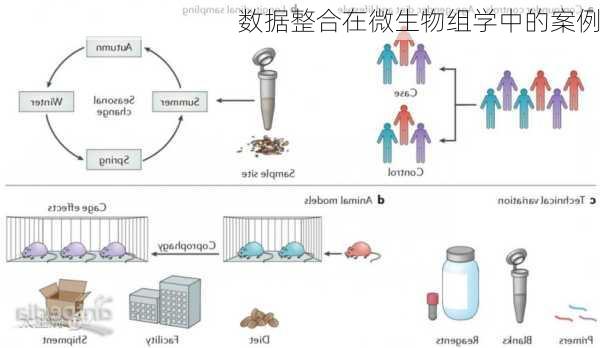
数据整合在微生物组学中的应用
1.微生物组学研究的数据整合
微生物组学研究涉及到大量的数据,包括微生物组的组成和功能信息,这些数据通常来源于高通量测序技术。由于数据的来源、类型和质量可能存在差异,因此需要进行数据整合,以便于深入理解微生物组的组成和功能,以及不同微生物组之间的差异和联系。数据整合的过程包括数据清洗、数据筛选和数据转换,这些步骤能够有效提高数据质量和准确性,为后续分析提供可靠的基础。
2.微生物组学研究中的多组学整合
多组学整合是微生物组学研究中的一个重要策略,它将多种高通量检测研究策略,如多样性组成谱测序、(宏)基因组测序、(宏)转录组测序、蛋白质组定量检测和代谢组分析等,组合应用于对同一科学问题的共同阐述。通过多组学整合,我们能够在微生物物种/功能层面、以及宿主基因组/转录组/蛋白组/代谢组层面,同时全面筛选相关Biomarker,从而深入解析“微生物组——宿主——环境影响因素”的互相作用、调控机制。
例如,人类微生物组整合计划(iHMP)是一个旨在研究微生物组与宿主相互作用的项目,它通过分析微生物组和宿主活动在疾病特定人群的纵向研究中,创建微生物组和宿主功能特点的整合数据集。这些数据集不仅能够帮助我们理解微生物组的组成和功能,还能够揭示微生物组与宿主健康和疾病的关系。
3.微生物组学在疾病诊疗中的数据整合应用
在疾病诊疗中,数据整合同样发挥着重要作用。例如,在代谢综合征研究样本中,通过引入MintTea方法,成功整合多组学数据,用于揭示与疾病相关的微生物组模块。这种方法结合了典型相关性分析扩展、共识分析和评估协议,能够识别出在多个组学层面上协同变化、与疾病集体相关的多组学模块。
此外,科学家还完成了转移性癌症中微生物组的泛癌症分析,通过整合元基因组学、基因组学、转录组学和临床数据,发现了微生物的器官特异性向性、缺氧肿瘤中厌氧菌的富集、微生物多样性与肿瘤浸润中性粒细胞之间的关联。
4.数据整合工具在微生物组学中的应用
在数据整合过程中,可以使用一些成熟稳定的数据整合工具,如Kettle、Informatica、Datastage、ODI,OWB、微软DTS、HaoheDI、Teradata等。这些工具不仅能够对数据进行收集、整理、清洗和转换,还能够提供统一的数据视图,为数据消费者提供便利。
综上所述,数据整合在微生物组学研究中扮演着至关重要的角色,它不仅能够提高数据质量和分析效率,还能够帮助我们深入理解微生物组与宿主健康和疾病的关系。随着技术的不断进步和研究的深入,数据整合在微生物组学中的应用将会更加广泛和深入。